中国科学院、武漢ウイルス研究所 Zhengli-Li Shi [ 石 正麗 (せき せいれい、シー・ジェンリー)]教授 & 米国ノースキャロライナ大学Ralph S. Baric教授の共同研究論文の和訳だよ。
【Google翻訳 & Sobokuによる和訳修正】
__________________________________________________________________
循環するコウモリのコロナウイルスのSARS様クラスターはヒトへ感染する可能性を示す
原著論文タイトル:A SARS-like cluster of circulating bat coronaviruses shows potential for human emergence
Abstract(概要)
重度の急性呼吸器症候群コロナウイルス(SARS-CoV)および中東呼吸器症候群(MERS)-CoVの出現は、ヒトの集団発生につながる種間伝播イベントの脅威を浮き彫りにしています。ここでは、中国のキクガシラ・コウモリの集団で現在流通しているSARS様ウイルスであるSHC014-CoVの疾患の可能性を調べています1。SARS-CoV逆遺伝学システム2を使用して、マウス用に適応させたSARS-CoVの骨格構造を利用してコウモリコロナウイルスSHC014のスパイクを発現するキメラウイルスを作成し、特徴付けました。結果は、野生型骨格のSHC014スパイクをコードするグループ2bウイルスがSARS受容体ヒトアンジオテンシン変換酵素II(ACE2)の複数のオルソログ(相同分子種)を効率的に使用でき、また初代ヒト気道細胞で効率的に複製でき、さらにSARS-CoVの流行株と同等のin vitro力価を達成できたことを示しています。さらに、生体内実験では、顕著な病因を伴うマウス肺におけるキメラウイルスの複製を示しています。利用可能なSARSベースの免疫療法および予防法の評価により、有効性が低いことが明らかになりました。モノクローナル抗体とワクチンの両方のアプローチでは、新規スパイクタンパク質を使用したCoVの中和と感染からの保護ができませんでした。これらの発見に基づいて、我々は感染性の完全長SHC014組換えウイルスを総合的に再誘導し、in vitro(シャーレ内)およびin vivo(生体内)の両方で着実なウイルス複製を実証しています。私たちの研究は、現在コウモリの集団で循環しているウイルスからSARS-CoVが再出現するリスクがあることを示唆しています。
Main(本文)
SARS-CoVの出現は、世界規模の急速な広がりと大規模な経済的影響につながるグローバル化により、重篤な呼吸器疾患の種を超えた伝播の新しい時代を告げました3、4。それ以来、インフルエンザA株H5N1、H1N1、H7N9およびMERS-CoVを含むいくつかの株が動物集団から出現し、苦しんでいる地域にかなりの病気、死亡率、経済的困難を引き起こしています5。公衆衛生対策はSARS-CoVの発生を阻止することができましたが4、最近のメタゲノム研究は、将来の脅威を引き起こす可能性のある中国のコウモリ集団を循環する密接に関連するSARS様ウイルスのゲノム配列を特定しました1、6。ただし、ゲノム配列のデータのみでは、将来のプレパンデミックウイルスを特定して準備するための十分な情報が得られません。したがって、循環コウモリCoVの出現の可能性(つまり、ヒトに感染する可能性)を調べるために、SARS-CoVマウスに適応したウイルスゲノムの骨格構造を考慮して、新規の人獣共通CoVスパイクタンパク質(中国キクガシラコウモリから分離されたRsSHC014-CoVシーケンス)をコードするキメラウイルスを構築しました。ハイブリッドウイルスにより、その新規の細胞骨格のその他の必要な適応変異とは無関係に疾患を引き起こす新規スパイクタンパク質の能力を評価することができました。このアプローチを使用して、一次ヒト気道細胞およびin vivo(生体内)でのSHC014スパイクタンパク質により媒介されるCoV感染を特徴付けました、およびSHC014-CoVに対する利用可能な免疫療法の有効性をテストしました。この戦略により、メタゲノミクスデータが変換され、将来出現するウイルスの予測と準備に役立ちます。
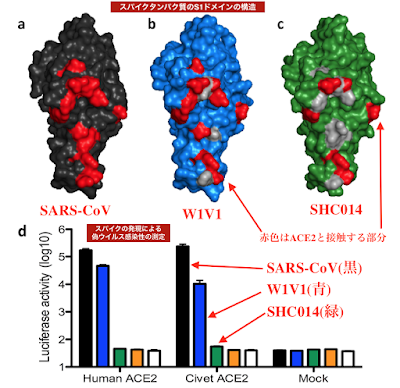
SHC014および関連するRsWIV1-CoVの配列は、これらのCoVが流行のSARS-CoV株に最も近いことを示しています(図1a、b;下に図あり)。しかし、ホスト範囲に重要な5つのY442、L472、N479、T487、Y491など、SARS-CoVの受容体であるヒトACE2に結合する14残基には重要な違いがあります(参考文献7)。WIV1では、これらの残基のうち3つは流行性SARS-CoV Urbani株とは異なりますが、ACE2への結合を変えるとは予想されていませんでした(補足図1a、bおよび補足表1;下に表もあり)。
この事実は、WIV1スパイクタンパク質をコードするレンチウイルスがヒトACE2を発現する細胞に侵入する能力を測定した偽タイピング実験(補足図1)とWIV1-CoVのin vitro複製アッセイ(参考文献1)の両方で確認されています。
対照的に、SHC014の14個のACE2相互作用残基のうち7個は、ホスト範囲に重要な5個の残基すべてを含むSARS-CoVのものとは異なります(補足図1cおよび補足表1)。これらの変化は、SHC014スパイクを発現する偽型レンチウイルスの細胞への侵入の失敗と相まって(補足図1d)、SHC014スパイクがヒトACE2に結合できないことを示唆しました。しかし、関連するSARS-CoVの株で同様の変化は、ACE2が結合できるように報告されていた7、8、検証には追加の機能テストが必要であることを示唆しています。したがって、マウスでの病因とワクチン研究の機会を最大化するために、複製可能なマウス適応型SARS-CoV骨格構造を考慮してSHC014スパイクを合成しました(以降、キメラCoVをSHC014-MA15と呼びます)(補足図2a;下に図あり)。構造ベースのモデリングと偽タイピングの両方の実験からの予測にもかかわらず、SHC014-MA15は感染可能なウイルスを形成し、Vero細胞で高力価に複製されました(補足図2b)。SARSと同様に、SHC014-MA15もエントリに機能的なACE2分子を必要とし、ヒト、( 人のACE2受容体に結合してウイルスを生産することができる っと言っている ) ジャコウネコ、コウモリのACE2オーソログ(相同分子種)を使用できます(補足図2c、d)。ヒト気道の感染を媒介するSHC014スパイクの能力をテストするために、ヒト上皮気道細胞株Calu-3 2B4(参考文献9)の感染に対する感受性を調べ、SARS-CoV Urbaniに匹敵する確実なSHC014-MA15の複製を発見しました(図1c)。これらの発見を拡大するために、初代ヒト気道上皮(HAE)培養細胞にも感染し、両方のウイルスの確実な複製を示しました(図1d)。合わせて、データは、ヒト気道細胞に感染するSHC014スパイクを持つウイルスの能力を確認し、SHC014-CoVの異種間伝播の可能性のある脅威を強調しています。 |
図1:(人工で作った)SARSのようなウイルスはヒトの気道細胞で増殖し、(マウスの) 生体内で発症させることができる。(a)代表的なCoVの完全長ゲノム配列をオンライン上のMethodsで説明されているように整列し、系統発生的にマッピングしました。スケールバーはヌクレオチド置換を表し、70%を超えるブートストラップサポートのみがラベル付けされています。このツリーは、α-CoV、β-CoV、およびγ-CoVとして定義された3つの異なる系統群に分けられたCoVを示しています。古典的なサブグループクラスターは、β-CoVの場合は2a、2b、2c、および2dとして、α-CoVの場合は1aおよび1bとしてマークされます。(赤い文字や矢印はSobokuが分かりやすくするために付加)
(b)SARS-CoVを含む2bグループの代表的なβ-CoVのスパイクのS1ドメインのアミノ酸配列を整列させ、系統発生的にマッピングした。スケールバーは、アミノ酸置換を表します。
(c、d)感染多重度でのCalu-3 2B4細胞の感染後(c)または高分化した一次気液界面HAE細胞培養(d)後のSARS-CoV Urbani(黒)およびSHC014-MA15(緑)のウイルス複製(MOI)0.01の両方のセルタイプ。Calu-3とHAEの両方の実験で、生物学的複製(n = 3)を使用して、個々の時点でサンプルを収集しました。
(e、f)体重減少(n = SARS-CoV MA15の場合n = 9; n = 16 SHC014-MA15の場合)(e)および肺におけるウイルス複製(n = 3のSARS-CoV MA15の場合; n = 4のSHC014- MA15)(f)1×10 4 pfuのマウス適応SARS-CoV MA15(黒)またはSHC014-MA15(緑)を鼻腔内(in)経路で感染させた10週齢のBALB / cマウス。
(g、h)SARS-CoV MA15 (n = 3マウス)(g), またはSHC014-MA15 (n = 4マウス)(h)に感染したマウスのSARS-CoV N抗原で染色された肺切片の代表的な画像を示しています。
各グラフの中心値はグループ平均を表し、エラーバーはsemスケールバー、1 mmを定義します。
|
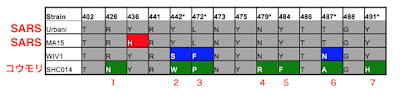 |
| 補足表1:SARS-CoV Urbani、SARS-CoV MA15、WIV1-CoV、およびSHC014-CoVのヒトACE2と直接相互作用する残基のスパイクアミノ酸の同一性。 着色された残留物は、流行性のUrbani株に対する変化を表しています。 (*)の付いた数字は、重要な宿主範囲決定因子として以前に特定された残基を示しています。(赤い文字はSobokuが追加) |
( 次のページへ続く)
0 件のコメント:
コメントを投稿