Soboku: インドの研究者による論文は、HIVの配列についてたくさんの批判を受けたが、論文は配列についてのみ言っているわけではない。
他に4つの特徴をこの論文では言及しているので、そのポイントを以下に列挙した、
また今はすでに取り下げられた論文の日本文を、その下に記録しておく。
______________________________
配列の類似性(HIVの一部に似ている)以外の4つのポイント
・4つの挿入のうち、最初の3つがHIVがgp120に類似している。
・gp120とは、CD4 Tリンパ球(CD4細胞)などの宿主細胞上のCD4受容体に結合し、HIVがウイルス膜を宿主細胞膜と融合させて宿主細胞に入るステップで重要である。
・3つがHIV・gp120に類似している部分は、2019-nCoVの立体構造予測をすると、距離が非常に近く、受容体結合部位の重要なコンポーネントを構成している可能性が高い。
・4つの挿入物すべてが約10のpI値を持ち、ウイルスとホストの相互作用を促進する可能性がある。
______________________________
Uncanny similarity of unique inserts in the 2019-nCoV spike protein to HIV-1 gp120 and Gag
論文タイトル(訳):2019-nCoVスパイクタンパク質とHIV-1のgp120およびGagのユニークなインサートの不思議な類似性
Prashant Pradhan$1,2, Ashutosh Kumar Pandey$1, Akhilesh Mishra$1, Parul Gupta1, Praveen Kumar Tripathi1, Manoj Balakrishnan Menon1, James Gomes1, Perumal Vivekanandan*1and Bishwajit Kundu*1
1Kusuma School of biological sciences, Indian institute of technology, New Delhi-110016, India. 2Acharya Narendra Dev College, University of Delhi, New Delhi-110019, India
$Equal contribution
$Equal contribution
Abstract:(概要)
現在、2019年の新規コロナウイルス(2019- nCoV)による大流行を目の当たりにしています。 2019-nCoVの進化はとらえどころのないままです。 2019-nCoVに固有であり、他のコロナウイルスには存在しないスパイク糖タンパク質(S)に4つの挿入が見つかりました。 重要なことに、4つのインサートすべてのアミノ酸残基は、HIV-1 gp120またはHIV-1 Gagのアミノ酸残基と同一または類似しています。興味深いことに、一次アミノ酸配列でインサートが連続していないにもかかわらず、2019-nCoVの3Dモデリングは、それらが受容体の結合部位を構成するようにまとまっていることを示しています。 HIV-1の重要な構造タンパク質のアミノ酸残基と同一性/類似性を有する2019-nCoVの4つのユニークなインサートの発見は、自然界では偶然ではありません。 この研究は、2019-nCoVに関する未知の洞察を提供し、このウイルスの診断と重要な意味を持つこのウイルスの進化と病原性に光を当てます。
Introduction(前書き)
コロナウイルス(CoV)は、動物や人間に感染する一本鎖のプラスセンスRNAウイルスです。これらは、宿主特異性に基づいて、4つの属に分類されます:アルファコロナウイルス、ベータコロナウイルス、デルタコロナウイルス、およびガンマコロナウイルス(Snijder et al。、2006)。 229EおよびNL63(アルファコロナウイルス属)、OC43、HKU1、MERSおよびSARS(ベータコロナウイルス属)を含む7種類の既知のCoVがあります。 229E、NL63、OC43、およびHKU1は一般にヒトに感染しますが、2002年と2012年のSARSとMERSのアウトブレイクは、それぞれウイルスが動物からヒトにクロスオーバーして重大な死亡を引き起こしたときに発生しました(J. Chan et al。、nd; JFW Chan et al。、2015)。 2019年12月、中国の武漢からコロナウイルスの別の発生が報告され、これも動物からヒトに感染しました。この新しいウイルスは、世界保健機関(WHO)によって一時的に2019-novel Coronavirus(2019-nCoV)と呼ばれています(J. F.- W. Chan et al。、2020; Zhu et al。、2020)。 2019-nCoVの起源についていくつかの仮説がありますが、この進行中の発生の原因はとらえどころのないままです。
2019-nCoVの伝播パターンは、ウイルスに感染した人との身体またはエアロゾルの接触を含む、以前のアウトブレイクで文書化された伝播パターンに類似しています。軽度から重度の症例、および感染による死亡が武漢から報告されています。このアウトブレイクは、フランス、オーストラリア、アメリカなど、遠く離れた国々に急速に広がりました。中国内外の症例数は急増しています。私たちの現在の理解は、ウイルスのゲノム配列と控えめな疫学的および臨床データに限定されています。利用可能な2019-nCoVシーケンスの包括的な分析は、進行中の大発生を管理するための現在の理解を深めるのに役立つ重要な手がかりを提供する可能性があります。
コルナナウイルスのスパイク糖タンパク質(S)は、2つのサブユニット(S1およびS2)に切断されます。 S1サブユニットは受容体の結合を助け、S2サブユニットは膜融合を促進します(Bosch et al。、2003; Li、2016)。コロノウイルスのスパイク糖タンパク質は、組織の向性と宿主域の重要な決定因子です。さらに、スパイク糖タンパク質はワクチン開発の重要なターゲットです(Du et al。、2013)。このため、スパイクタンパク質はコロナウイルスの中で最も広範囲に研究されています。したがって、計算ツールを使用して、2019-nCoVのスパイク糖タンパク質を調査し、その進化、新規機能シーケンス、および構造的機能を理解しようとしました。
Methodology(方法論)
核酸およびタンパク質配列の検索とアライメント
NCBIウイルスゲノムデータベース(https://www.ncbi.nlm.nih.gov/)から利用可能なすべてのコロナウイルス配列(n = 55)を取得し、GISAIDを使用しました(Elbe&Buckland-Merrett、2017)[https: //www.gisaid.org/] 2020年1月27日現在の2019- nCoVのすべての利用可能な完全長配列(n = 28)を取得します。MUSCLEソフトウェアを使用して、すべてのコロナウイルスゲノムの複数配列アライメントを実行しました(Edgar、2004)近隣参加方法に基づいています。 55個のコロナウイルスゲノムのうち、すべてのカテゴリの32個の代表的なゲノムが、MEGAXソフトウェアを使用した系統樹の開発に使用されました(Kumar et al。、2018)。最も近い親relativeはSARS CoVであることがわかった。 SARS CoVと2019-nCoVの糖タンパク質領域は、Multalinソフトウェア(Corpet、1988)を使用して整列および可視化されました。同定されたアミノ酸およびヌクレオチド配列は、BLASTpおよびBLASTnを使用して、ウイルスゲノムデータベース全体と整列されました。 2019-nCoVゲノムの28の臨床変異体におけるヌクレオチドおよびアミノ酸モチーフの保存は、MEGAXソフトウェアを使用して複数の配列アラインメントを実行することによって提示されました。 2019-nCoV糖タンパク質の3次元構造は、SWISS-MODELオンラインサーバーを使用して生成され(Biasini et al。、2014)、PyMolを使用して構造がマークおよび視覚化されました(DeLano、2002)。
Results(結果)
2019-nCoVスパイクタンパク質の新規挿入物とHIV-1 gp120およびGagとの不思議な類似性
全長コロナウイルスの系統樹は、2019-nCoVがSARS CoVと密接に関連していることを示唆しています[図1]。さらに、他の最近の研究により、2019-nCoVはSARS CoVにリンクされています。したがって、2019-nCoVのスパイク糖タンパク質配列をSARS CoVのスパイク糖タンパク質配列と比較しました(NCBIアクセッション番号:AY390556.1)。配列アラインメントを注意深く調べると、2019- nCoVスパイク糖タンパク質には4つの挿入が含まれていることがわかりました[図2]。
NCBI refseq(ncbi.nlm.nih.gov)の利用可能なすべてのコロナウイルス(n = 55)[表S.File1を参照]のスパイク糖タンパク質アミノ酸配列には、2019-nCoVの1つの配列が含まれます[図S1] 。これらの4つの挿入[挿入1、2、3、および4]は2019-nCoVに固有であり、分析された他のコロナウイルスには存在しないことがわかりました。中国からの別のグループは、コロナウイルスのより少ないスパイク糖タンパク質配列を比較する3つの挿入を文書化しました。中国の別のグループは、コロナウイルスのより少ないスパイク糖タンパク質配列を比較する3つの挿入を記録しました(Zhou et al。、2020)。
 |
| 図2:2019-nCoVとSARSのスパイクタンパク質間の複数の配列アライメント。2019-nCoV(Wuhan-HU-1、Accession NC_045512)およびSARS CoV(GZ02、Accession AY390556)のスパイクタンパク質の配列は、MultiAlinソフトウェアを使用してアライメントされました。違いのあるサイトはボックスで強調表示されます。 |
次に、これらの挿入物の存在について、2020年1月27日の時点で、GISAID(Elbe&Buckland-Merrett、2017)で2019-nCoVの利用可能なすべての完全長シーケンス(n = 28)を分析しました。これらの配列のほとんどには注釈が付けられていないため、BLASTpを使用して、利用可能なすべての2019-nCoV配列のスパイク糖タンパク質のヌクレオチド配列を比較しました。興味深いことに、分析されたすべての利用可能な2019-nCoV配列で4つの挿入すべてが完全に(100%)保存されていました[図S2、図S3]。
次に、整列したゲノムを翻訳し、これらの挿入物がホストとしてのBatの2019-nCoVウイルスを除くすべてのWuhan 2019- nCoVウイルスに存在することを発見しました[図S4]。 2019-nCoVに固有の高度に保存された4つのインサートに興味をそそられ、その起源を理解したいと考えました。この目的のために、すべてのウイルスゲノムに対するクエリとして各挿入物と2019-nCoVローカルアラインメントを使用し、100%のシーケンスカバレッジでヒットと見なしました。驚くべきことに、4つの挿入物のそれぞれは、ヒト免疫不全ウイルス1(HIV-1)タンパク質の短いセグメントと整列しています。 2019-nCoVの挿入物のアミノ酸位置と、HIV-1 gp120およびHIV-1 Gagの対応する残基を表1に示します。最初の3つの挿入物(挿入物1、2および3)はアミノ酸の短いセグメントに整列しています。 HIV-1 gp120の残留物。挿入物4はHIV-1 Gagに整列しました。 2019-nCoVのスパイク糖タンパク質の挿入1(6アミノ酸残基)および挿入2(6アミノ酸残基)は、HIV-1 gp120にマッピングされた残基と100%同一です。 2019- nCoVの挿入3(12アミノ酸残基)は、ギャップのあるHIV-1 gp120にマップされます[表1を参照]。インサート4(8アミノ酸残基)は、ギャップのあるHIV-1 Gagにマップされます。
4つのインサートは2019-nCoVのスパイク糖タンパク質のアミノ酸の不連続な短いストレッチを表していますが、3つすべてが(すべての注釈付きウイルスタンパク質の中で)HIV-1 gp120およびHIV-1 Gagとアミノ酸の同一性または類似性を共有しているという事実これは偶然の発見ではないことを示唆しています。言い換えれば、無関係なタンパク質中の連続した6~12個の連続したアミノ酸残基の偶然の一致を散発的に期待するかもしれません。ただし、2019-nCoVスパイク糖タンパク質の4つすべての挿入物が、無関係なウイルス(HIV-1)の2つの重要な構造タンパク質と偶然に一致する可能性は低いです。
HIV-1にマッピングされた2019-nCoVスパイク糖タンパク質のインサート1、2、3のアミノ酸残基は、それぞれgp120のV4、V5、V1ドメインの一部でした[表1]。 2019- nCoVインサートはHIV-1の可変領域にマッピングされているため、HIV-1 gp120では広範に分布しませんでしたが、主にアジアおよびアフリカのHIV-1 [S.File1を参照]の選択された配列に限定されました。
HIV-1 Gagタンパク質は、ウイルスと負に帯電した宿主表面との相互作用を可能にし(Murakami、2008)、Gagタンパク質の高い正電荷は宿主ウイルス相互作用の重要な特徴です。 2019-nCoVの4つの挿入物のそれぞれのpI値とHIV-1タンパク質のアミノ酸残基の対応するストレッチを分析すると、a)分析された各ペアでpI値が非常に類似していることがわかりました。b)これらのpI値のほとんど10±2 [表1参照]。注目すべきは、インサート3と4のギャップにもかかわらず、pI値は同等であったことです。 4つのインサートすべてのpI値のこの均一性は、さらに調査する価値があります。
これらの4つのインサートはいずれも他のコロナウイルスには存在しないため、これらのインサートをコードするゲノム領域は、2019-nCoVと他のコロナウイルスを区別できるプライマーを設計するための理想的な候補となります。
 |
| 表1:HIV-1の2019-nCoVおよびgp120タンパク質の配列とタンパク質の一次配列の位置。すべてのインサートには、高密度の正に帯電した残留物があります。挿入物3および4の削除されたフラグメントは、正電荷対表面積比を増加させます。* Suppを参照してください。アクセッション番号の表1 |
新規インサートは2019-nCoVの受容体結合部位の一部です
構造的洞察を取得し、2019-nCoV糖タンパク質におけるこれらの挿入の役割を理解するために、SARSスパイク糖タンパク質(PDB:6ACD.1.A)の利用可能な構造に基づいてその構造をモデル化しました。モデル化された構造の比較により、インサート1、2、3はタンパク質の一次配列の不連続な位置にありますが、折り畳まれて宿主受容体を認識する糖タンパク質結合部位の一部を構成していることがわかります(Kirchdoerfer et al。、2016) (図3)。インサート1は2019-nCoVスパイク糖タンパク質のS1サブユニットのNTD(N末端ドメイン)に対応し、インサート2および3はCTD(C末端ドメイン)に対応します。インサート4は、S1サブユニットのSD1(サブドメイン1)とSD2(サブドメイン2)の接合部にあります(Ou et al。、2017)。これらの挿入は、ウイルスと宿主の相互作用を促進または強化する親水性ループをタンパク質構造に形成することにより、糖タンパク質結合部位にさらなる柔軟性を提供すると推測します。
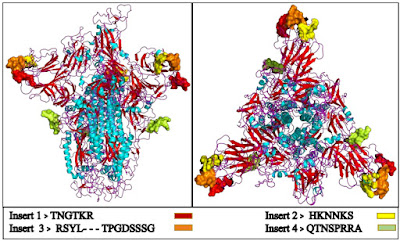 |
| 図3. 2019-nCoVウイルスのモデル化されたホモ三量体スパイク糖タンパク質。HIVエンベロープタンパク質からの挿入物は、タンパク質の結合部位に存在する色付きのビーズで示されています。 |
2019-nCoVの進化分析
2019-nCoVは、人間に感染した動物源に由来するコロナウイルスのバリアントであると推測されています。宿主の特異性の変化を考慮して、ウイルスのスパイク糖タンパク質(Sタンパク質)の配列を調べることにしました。 Sタンパク質は、宿主の認識と付着においてウイルスを助ける表面タンパク質です。したがって、これらのタンパク質の変化は、ウイルスの宿主特異性の変化として反映されます。 2019-nCoVのSタンパク質遺伝子の変化と構造再構成におけるその結果を知るために、他のすべてのウイルスに関して2019-nCoVのシリコ分析を行いました。 2019-nCoV、Bat-SARS-Like、SARS-GZ02、MERSのSタンパク質アミノ酸配列間の複数の配列アラインメントにより、Sタンパク質がSARS-GZ02に最も近い有意な多様性で進化したことが明らかになりました(図1)。
2019-nCoVのスパイクタンパク質領域への挿入
2019-nCoVのSタンパク質はSARS GZ02と最も近い祖先を共有しているため、これら2つのウイルスのスパイクタンパク質をコードする配列をMultiAlinソフトウェアを使用して比較しました。 2019-nCoV-「GTNGTKR」(IS1)、「HKNNKS」(IS2)、「GDSSSG」(IS3)、および「QTNSPRRA」(IS4)のタンパク質に4つの新しい挿入が見つかりました(図2)。驚いたことに、これらの配列挿入はSARSのSタンパク質に存在しなかっただけでなく、コロナウイルス科の他のメンバーでも観察されませんでした(補足図)。これは、ウイルスがこのようなユニークな挿入を短時間で自然に獲得する可能性が極めて低いため、驚くべきことです。
挿入物はHIVと類似している
挿入は、最近の臨床分離株から入手可能な2019-nCoVウイルスのすべてのゲノム配列に存在することが観察されました(補足図1)。 2019-nCoVでのこれらの挿入のソースを知るために、すべてのウイルスゲノムでクエリとしてこれらの挿入を使用して、BLASTpでローカルアライメントが行われました。予想外に、すべての挿入がヒト免疫不全ウイルス-1(HIV-1)と一致しました。さらに分析したところ、2019-nCoVを含むHIV-1の整列配列は、表面糖タンパク質gp120(アミノ酸配列位置:404-409、462-467、136-150)およびGagタンパク質(366-384アミノ酸)(表1)。 HIVのGagタンパク質は、宿主膜結合、ウイルスのパッケージング、およびウイルス様粒子の形成に関与しています。 Gp120は、一次受容体CD4に結合することで宿主細胞を認識する際に重要な役割を果たします。
Discussion(討論)
2019-nCoVの現在の発生は、人間に感染する能力の徹底的な調査と理解を保証します。以前のコロナウイルスからこのウイルスへの宿主の選好に明確な変化があったことを念頭に置いて、我々は2019-nCoVと他のウイルス間のスパイクタンパク質の変化を研究しました。 2019-nCoVのSタンパク質には、その最も近い親族であるSARS CoVと比較して、4つの新しい挿入が見つかりました。最近の28の臨床分離株のゲノム配列は、これらの挿入をコードする配列がこれらすべての分離株間で保存されていることを示しました。これは、これらの挿入が2019-nCoVによって取得されたことが好ましいことを示しており、追加の生存と感染性の利点を提供します。さらに深く掘り下げると、これらの挿入はHIV-1に似ていることがわかりました。私たちの結果は、HIVのgp120とGagタンパク質と2019-nCoVスパイク糖タンパク質との驚くべき関係を強調しています。これらのタンパク質は、ウイルスが宿主細胞を特定してラッチするため、およびウイルスのアセンブリにとって重要です(Beniac et al。、2006)。表面タンパク質は宿主指向性の原因であるため、これらのタンパク質の変化は、ウイルスの宿主特異性の変化を意味します。中国からの報告によると、このウイルスはもともと動物ではなくヒトに感染することが知られていましたが、変異後、ヒトへの指向性も獲得したため、2019-nCoVの場合に宿主特異性が増加しました。
先に進むと、タンパク質構造の3Dモデリングは、これらの挿入が2019-nCoVの結合部位に存在することを示しました。 2019-nCoVスパイク糖タンパク質の結合ドメインにgp120モチーフが存在するため、これらのモチーフの挿入により、宿主細胞受容体に対する親和性が強化された可能性があります。さらに、この構造変化により、2019-nCoVが感染できる宿主細胞の範囲も拡大した可能性があります。私たちの知る限り、これらのモチーフの機能はHIVではまだ明確ではないため、調査する必要があります。ウイルス間での遺伝物質の交換はよく知られており、そのような重要な交換は、一見無関係なウイルスファミリー間の関係を調査するリスクと必要性を強調しています。
Conclusions(結論)
2019-nCoVのスパイク糖タンパク質の分析により、いくつかの興味深い発見が明らかになりました。まず、2019年までに報告された他のコロナウイルスには存在しない2019-nCoVスパイク糖タンパク質の4つのユニークな挿入物を特定しました。驚いたことに、NCBIデータベース内のすべての注釈付きウイルスタンパク質の中で、HIV-1 gp120およびGagのアミノ酸の短いセグメントにマッピングされた2019-nCoVの4つの挿入物すべて。 2019- nCoVスパイクタンパク質の新規挿入物とHIV-1 gp120およびGagのこの不思議な類似性は、偶然ではないでしょう。さらに、3Dモデリングは、2019-nCoVスパイク糖タンパク質の一次タンパク質配列で隣接していない少なくとも3つのユニークなインサートが収束して、受容体結合部位の重要なコンポーネントを構成することを示唆しています。注目に値するのは、4つの挿入物すべてが約10のpI値を持ち、ウイルスとホストの相互作用を促進する可能性があることです。まとめると、我々の発見は2019-nCoVの型破りな進化を示唆しており、さらなる調査が必要です。私たちの研究は、2019-nCoVの新しい進化的側面を強調し、このウイルスの病因と診断に影響を与えます。
引用(PDF書類):
https://www.biorxiv.org/content/10.1101/2020.01.30.927871v1.full.pdf
0 件のコメント:
コメントを投稿